Autor: Bernardo Bello Ortí
Estudiante de doctorado en microbiología. Subprograma Infecciones Bacterianas, línea H. parasuis (CReSA)

El estudio de las enfermedades causadas por infecciones bacterianas y víricas está cambiando. En los últimos años, técnicas como la secuenciación masiva de ADN están aportando una cantidad ingente de información que era impensable hace unas décadas. La aplicación de dicha técnica está siendo paulatinamente introducida en medicina humana; prevemos, pues, que en un futuro cercano sea también útil en la medicina veterinaria.
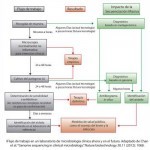
El diagnóstico es una de las áreas beneficiadas de esta revolución. Tomemos como ejemplo un brote infeccioso bacteriano; el procedimiento clásico de identificación del brote, la elección de la terapia apropiada y la gestión del mismo ha consistido y consiste en la recogida de muestras, inspección al microscopio y cultivo más determinación de género/especie por pruebas bioquímicas, o alternativamente diagnóstico por técnicas moleculares (PCR), determinación de la sensibilidad del patógeno a antibióticos y tipado del aislado cuando se estima oportuno y necesario. Por tanto, suele ser un proceso lento y, en ocasiones, tedioso. La secuenciación masiva es capaz de reducir este proceso de algunos días a menos de 24 horas. Sin embargo, su alto coste económico hace que las técnicas clásicas junto a las moleculares como la PCR o las de determinación de anticuerpos como ELISA dominen en la actualidad. Aunque técnicas como la PCR y la PCR a tiempo real son también muy rápidas, solamente pueden aplicarse a la detección de un numero limitado de especies bacterianas debido a las bases intrínsecas de la técnica. Otra variante, la metagenómica, consiste en la secuenciación masiva de ADN directamente de las muestras y comparación con bases de datos de patógenos. De este modo, la elección del antibiótico apropiado y genotipado del patógeno se realiza sin necesidad de cultivo. Sin embargo, esta nueva técnica no excluye a las anteriores sino que puede complementarse con ellas ya que el cultivo del patógeno puede facilitar el diagnóstico bioinformático.

Una vez se ha conseguido aislar la bacteria, la genómica, técnica basada en secuenciación de ADN de un cultivo puro abre nuevos campos de investigación como la detección de factores de virulencia, el genotipado, el diseño de medios de cultivo y el desarrollo de vacunas. Por otro lado, el genoma de este nuevo aislado puede ser comparado con otros ya disponibles (genómica comparativa) para ver las regiones comunes y específicas del aislado, genes de resistencia a antibióticos (resistoma) y elementos móviles (moviloma), comúnmente relacionados con cepas patógenas.

Aunque las aplicaciones de la genómica son muchas, no indican si estos genes están siendo o no usados por la bacteria. Usando la técnica de transcriptómica o secuenciación masiva de ARN se puede estudiar el metabolismo activo de la bacteria en un momento determinado, y así tener más conocimiento sobre la importancia de un determinado gen para una bacteria en el establecimiento de la infección. El siguiente nivel, la identificación de proteínas a escala masiva por técnicas como en MALDI-TOF (proteómica), va un paso mas allá dando ya evidencias claras de que un gen esta siendo usado por la bacteria en la infección (patofisiología). Aquí se amplían y afianzan los conocimientos para desarrollo de antibióticos, vacunas y desarrollo de test de diagnóstico (serología y anticuerpos monoclonales).
Dado que la mayoría de las bacterias no crecen en los medios de cultivos usados actualmente en los laboratorios, la secuenciación directa de ADN o ARN de muestras usando la técnica de metagenómica puede responder preguntas complejas como cuál es el metabolismo de una comunidad de microorganismos, la composición de microbiota, la estructura de una comunidad y su evolución, solapando con la genómica en la identificación de patógenos y resistencias a antibióticos.
Es de esperar que a medida que los costes de secuenciación sean mas accesibles, la genómica y metagenómica empiecen a tomar más importancia en el control de enfermedades infecciosas, complementando así a la microbiología clásica, pero reemplazándola además en algunos ámbitos como la identificación de patógenos o la resistencia a antibióticos.